MiXCR
免疫レパトア解析ソフトウェア
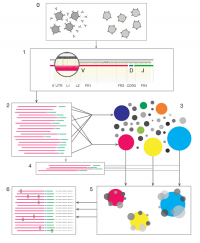
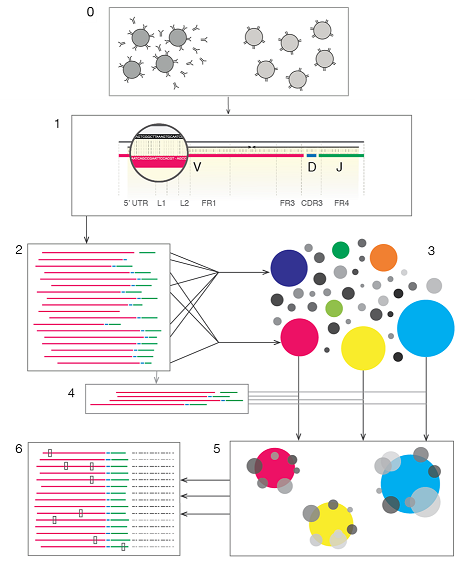
次世代シーケンサーの登場によって、T細胞やB細胞の表面に発現するT細胞受容体 (TCR) やB細胞受容体 (BCR) の遺伝子配列の多様性を網羅的に解析できるようになりました。 TCR/BCRレパトア (レパートリー) 解析は免疫状態の変化を定量化することで、医薬品や免疫療法の有効性評価、白血病や悪性リンパ腫の診断、骨髄移植後の免疫機能回復の評価、CAR-T細胞療法研究、コロナウイルス感染症研究、ネオアンチゲン免疫療法研究等にも応用することができる技術です。
MiLaboratories社のMiXCRは、TCR/BCRレパトア解析におけるゴールドスタンダードとされ、ユーザーはサンプルから迅速かつ正確にあらゆるレパートリーを解析することが可能です。
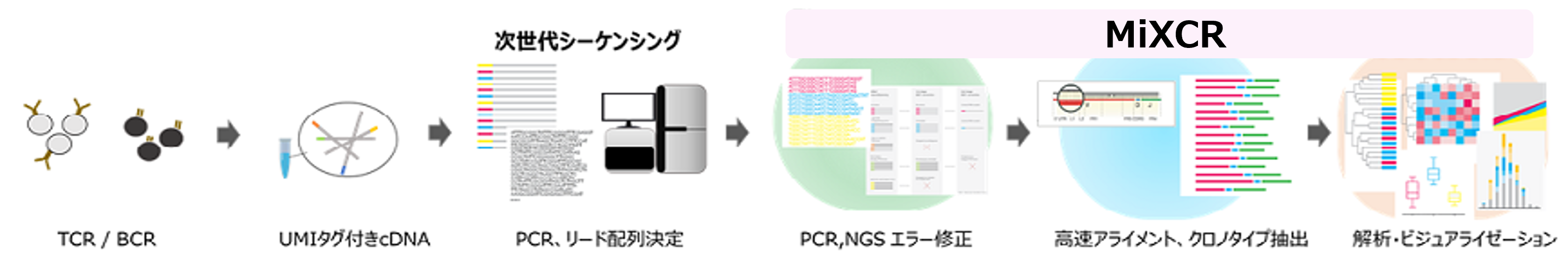
~ MiXCRのレパトア解析フロー ~
MiXCRについて
MiXCRの特徴
MiXCR は、T 細胞または B 細胞受容体におけるレパトアシーケンスの生データを迅速かつ正確に解析するためのソフトウェアです。あらゆる種類のシーケンスデータで動作します。
- UMIを含むまたは含まないバルクレパトアシーケンスデータ
- 10x Genomixプロトコルを含むシングルセルシーケンスデータ
- RNA-Seqや、ターゲットシーケンスの一部を含む可能性のあるフラグメント/ショットガンデータ
- TCR または BCR を含むその他のシーケンスデータ
強力なデータ解析ツールにより、複数の指標についてベクトルプロットや表形式の結果を得ることができます。主な機能は以下の通りです。
- メタデータの値でサンプルをグループ化し、グループ間でレパートリーの特徴を比較する機能
- 包括的なレパートリーの正規化とフィルタリング
- 適切な p 値調整による統計的有意性検定
- レパートリー重複解析
- ベクトルプロット出力 (.svg / .pdf)
- 表形式出力
その他の特徴は以下の通りです。
- 全長可変領域を含む任意の遺伝子特徴によるクロノタイプアセンブリ
- UMIまたはCellバーコードの有無にかかわらずPCR/シーケンスエラーを修正
- 偽陽性率ゼロで最大限の抽出を行う、堅牢な専用アライナーアルゴリズム
- あらゆるカスタムバーコードアーキテクチャ(UMI / Cell)をサポート
- ヒト、マウス、ラット、スパラックス、アルパカ、サルのデータをサポート
- IMGTリファレンスをサポート
- バーコードのエラー修正
- アダプタートリミング
- リードポジションが一定していない場合(ショットガンシーケンス、RNA-Seqなど)、断片化されたシーケンスリードのオーバーラップ部分を、CDR3を含む完全なコンティグにアセンブルしてCDR3を再構成するオプション
- 利用可能なデータから可能な限り長いTCR/IG配列を構築するためのコンティグアセンブリオプション(UMIまたはCellバーコードの有無は問わない)
- パイプラインのすべてのステップで提供される包括的な品質管理レポート
- データでカバーされていない領域をジャームラインから推定
- パイプライン全体を通して各生シーケンスリードの運命をバックトラックする機能
- クロノタイプとアラインメントに関する網羅的な出力情報
- すべての免疫学的関連領域(FR1、CDR1、…、CDR3など)の塩基配列およびアミノ酸配列
- 同定されたV、D、J、C遺伝子、ヌクレオチドおよびアミノ酸変異に関する包括的情報
- ヌクレオチドおよびアミノ酸変異に関する包括的情報
- 出力配列中のすべての免疫学的関連ポイントの位置
- その他の多くの有益情報
解析手順
MiXCRは以下の手順に沿って解析を行います。
- TCR/BCRの遺伝子配列データをセットします。
- シングル、ペアリードにおけるミスマッチ、挿入欠失を効率的に処理しアライメントを行います。
- 中間アライメント結果が抽出されます。
- 同一配列のものが同一クロノタイプとしてグルーピングされます。
- クオリティの低いシーケンスリードはさらなるマッピング用に収集されます。
- PCRとシーケンスエラーが修正されます。
- 最終的なクロノタイプがタブ区切りのファイルとして抽出されます。
MiXCRのユーザー
MiXCRは世界をリードする製薬企業10社のうち8社が以下の研究開発に使用しています。
- ワクチン開発
- 抗体探索
- がん免疫療法研究
学術界で広く採用され、査読付き科学論文で1000件以上引用されています。
稼働環境
MiXCRの稼働環境は以下となります。
- OS: Java実行環境が利用可能な Windows、Linux、MAC OS X(いずれのOSもバージョンは問いません)
- Java Runtime Environment: バージョン 8 以上
- メモリ: 1 – 16 GB (サンプルのクローン数に依存します)
文献情報
Bolotin D et al. MiXCR: software for comprehensive adaptive immunity profiling. Nature methods 12, no. 5 (2015): 380-381. doi:10.1038/nmeth.3364
Sharonov GV, Serebrovskaya EO, Yuzhakova DV, Britanova OV, Chudakov DM. B cells, plasma cells and antibody repertoires in the tumour microenvironment. Nat Rev Immunol.2020 Jan 27. doi: 10.1038/s41577-019-0257-x.
Li N, van Unen V, Abdelaal T, Guo N, Kasatskaya SA, Ladell K, McLaren JE, Egorov ES, Izraelson M, Chuva de Sousa Lopes SM, Höllt T, Britanova OV, Eggermont J, de Miranda NFCC, Chudakov DM, Price DA, Lelieveldt BPF, Koning F. Memory CD4+ T cells are generated in the human fetal intestine. Nature Immunology. 2019 Mar;20(3):301-312. doi: 10.1038/s41590-018-0294-9.
Plitas G, Konopacki C, Wu K, Bos PD, Morrow M, Putintseva EV, Chudakov DM, Rudensky AY. Regulatory T Cells Exhibit Distinct Features in Human Breast Cancer. Immunity. 2016 Nov 15;45(5):1122-1134. doi: 10.1016/j.immuni.2016.10.032.
Bolotin DA, Poslavsky S, Davydov AN, Frenkel FE, Fanchi L, Zolotareva OI, Hemmers S, Putintseva EV, Obraztsova AS, Shugay M, Ataullakhanov RI, Rudensky AY, Schumacher TN, Chudakov DM. Antigen receptor repertoire profiling from RNA-seq data. Nat Biotechnol. 2017 Oct 11;35(10):908-911. doi: 10.1038/nbt.3979.
Zvyagin IV, Tsvetkov VO, Chudakov DM, Shugay M. An overview of immunoinformatics approaches and databases linking T cell receptor repertoires to their antigen specificity. immunogenetics. 2020 Feb;72(1-2):77-84. doi: 10.1007/s00251-019-01139-4.
※記載の商品名等は各社の登録商標、または商品の場合があります。
