PEAKS
統合プロテオミクス解析ソフトウェア

BSI社のPEAKSはタンデム質量分析装置から得られたMS/MSピークリストから、de novoシーケンシングとデータベースサーチを行う、プロテオーム統合解析ソフトウェアです。
現在プロテオーム研究で主流となっているデータベースサーチ機能では、既知タンパク質のデータベースと照らし合わせることによって、サンプル中に含まれるタンパク質や、翻訳後修飾の解析を行うことが出来ます。
一方、de novoシーケンシング機能では、データベースに依存せずに、サンプル中に含まれるターゲットタンパク質のアミノ酸配列や、翻訳後修飾の解析を行うことが出来ます。データベースを必要としないde novoシーケンシングは、未知タンパク質やデータベースが整っていない種のタンパク質、生体内ペプチドや合成ペプチドなどの解析に有効です。
PEAKSは、de novoシーケンシング・データベースサーチ・翻訳語修飾解析・Mutation解析・糖鎖解析(Glycan Search)・定量解析(オプションプログラム:PEAKS Qを利用)を一つのソフトウェアで使用することが可能なので、研究者の様々なプロテオーム解析ニーズに対応が可能です。
PEAKSについて
PEAKSの主な機能
PEAKSでは、以下のフローに沿って解析を行います。
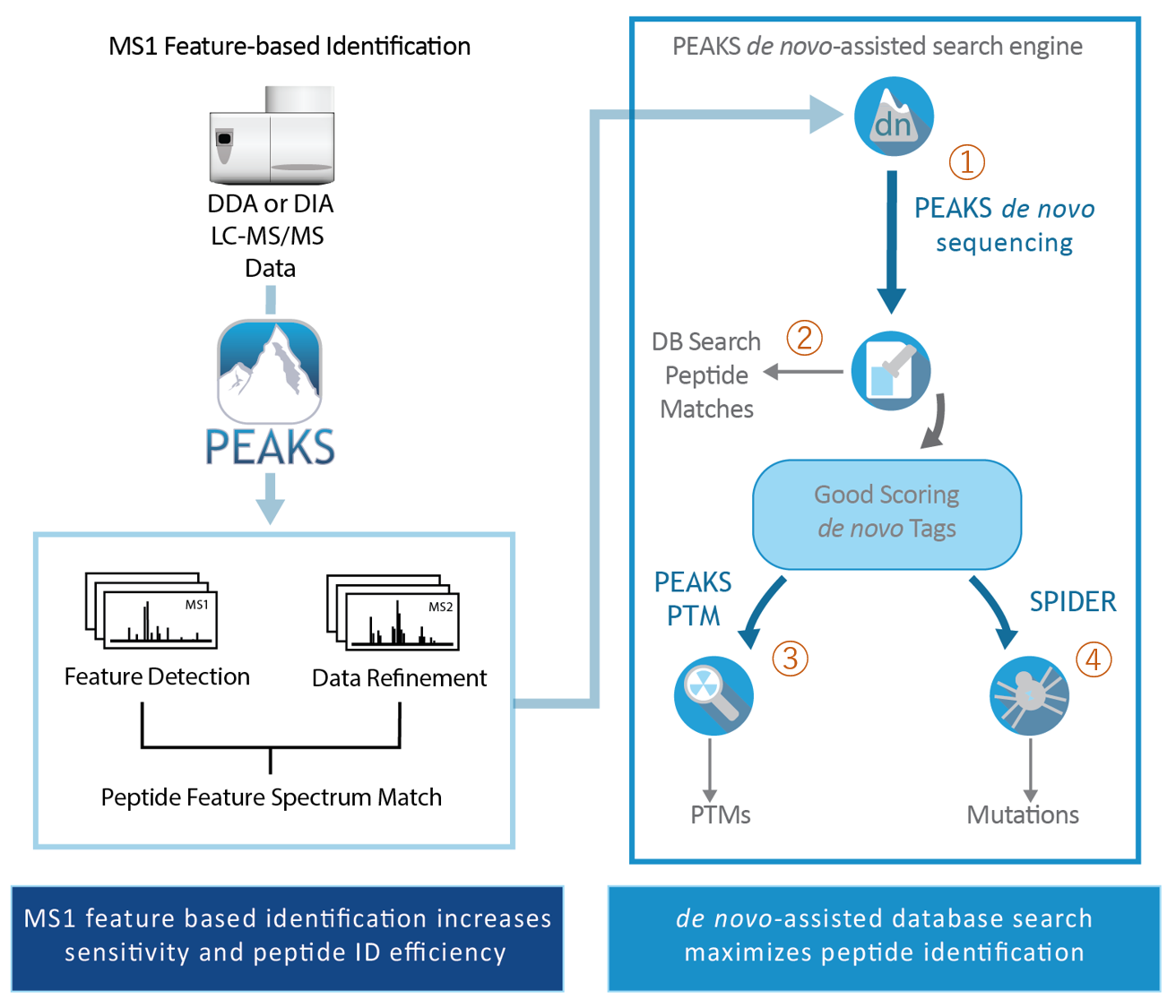
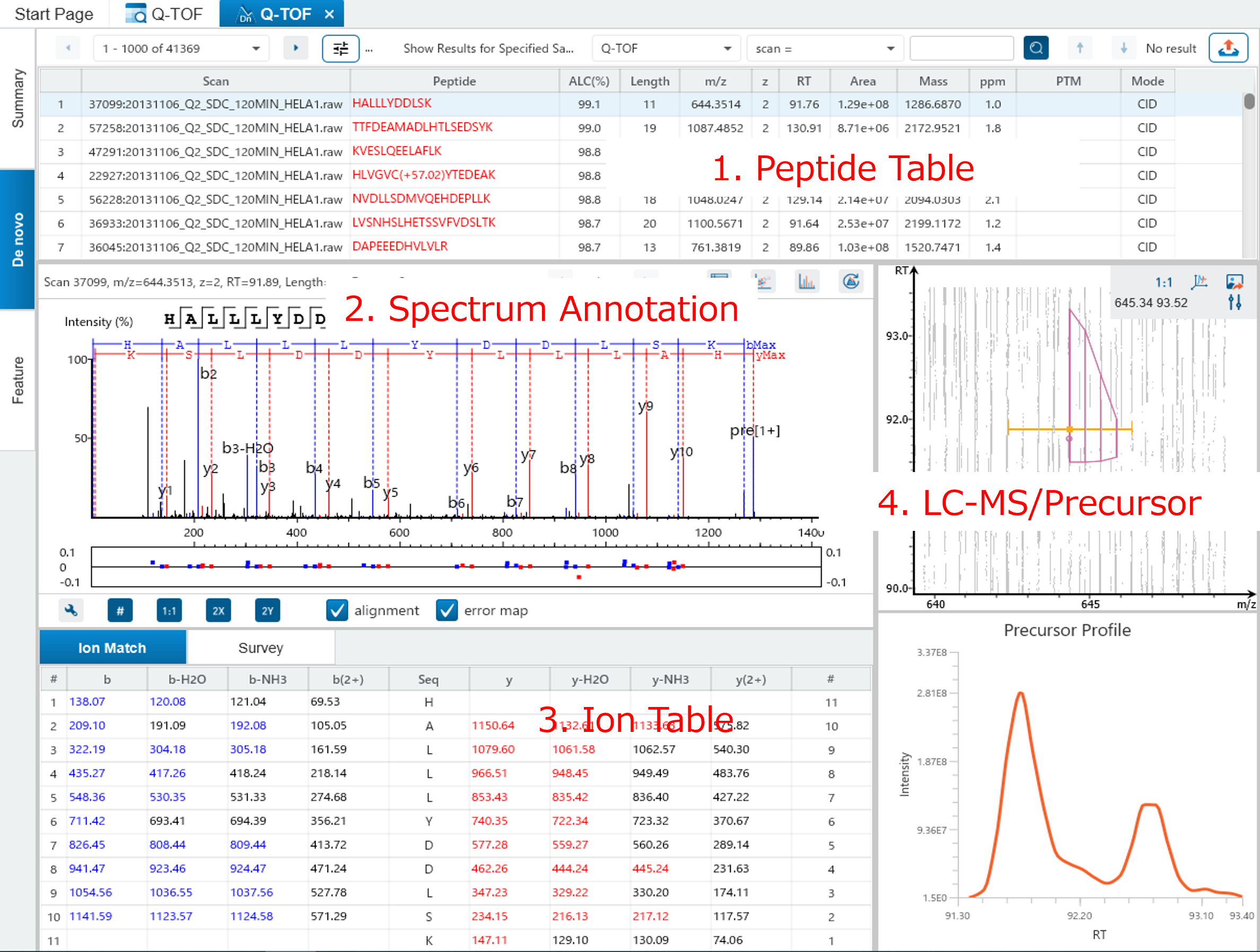
① de novoシーケンシング
de novoシーケンシングは、MS/MSから得られたイオンの質量をもとに、ペプチドのアミノ酸配列(翻訳後修飾含)を算出します。
データベースは全く使用しませんので、データベースに登録されていない配列の解析も可能です。
手動で行うと非常に面倒なde novoシーケンシングも、PEAKSではワンクリックでOKです。
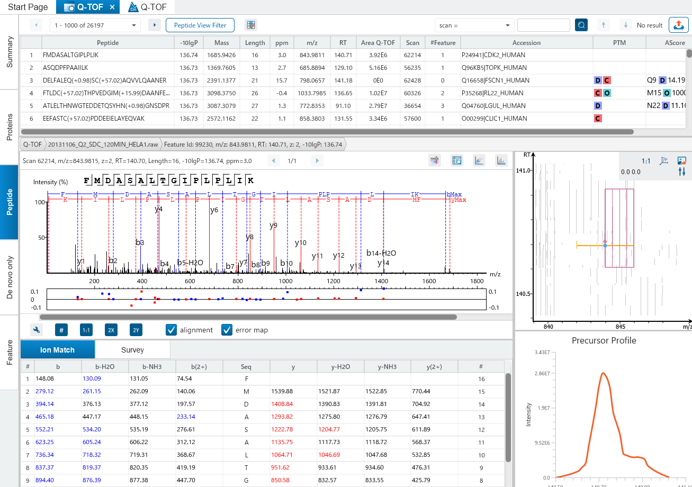
② データベースサーチ
既知タンパク質がターゲットの場合、データベースサーチ機能でタンパク/ペプチドの同定を行います。
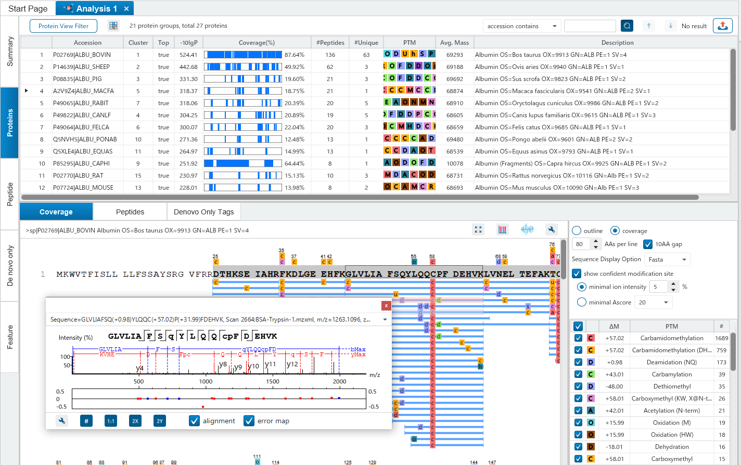
同定結果は、Summary・Protein・Peptide・Feature毎にタブ形式で見やすく設計され、また、グループタンパク(ファミリータンパク)がまとめて表示されるので、タンパク質としての検索スコアが非常に見やすくなっています。
更に未同定のMS/MSに対しては、de novno結果を参照し、一部配列がマッチしているスペクトルなどをチェックすることも可能です。
 ③ 翻訳後修飾 『PTM Finder』
③ 翻訳後修飾 『PTM Finder』
通常、DBサーチの時点では翻訳後修飾を多く設定することができず、そのために未設定のVariable修飾が原因で、未同定になる場合があります。
PEAKSのPTM Finder機能では、未同定のMS/MSかつ同定済タンパク質に対して、網羅的に翻訳後修飾をVariableで再解析を行うことができます。
これにより、全体のカバレージがアップします。
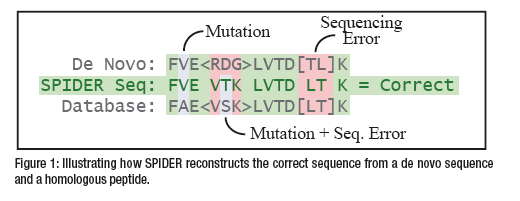 ④ Mutation解析 『SPIDER』
④ Mutation解析 『SPIDER』
データベースサーチや、網羅的な翻訳後修飾サーチでも同定されなかったMS/MSに対しては、ホモロジーサーチ機能を持つSPIDERを適用することで、Mutationを示唆することが可能です。
その他の機能
スペクトルライブラリサーチ『PEAKS Library Search』
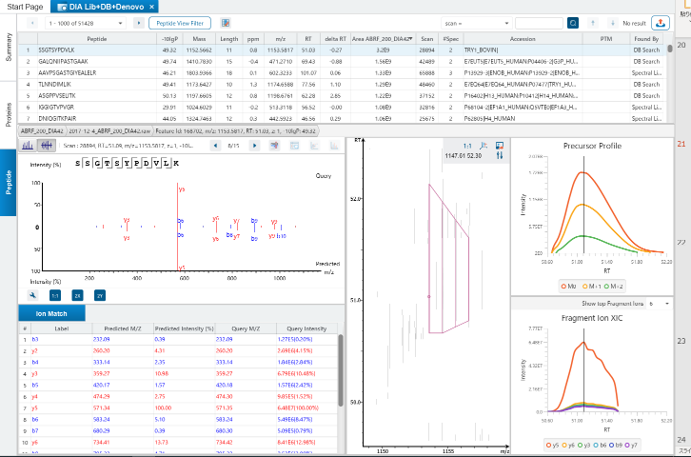
PEAKS Library Search では、ペプチドやタンパク質の同定のための通常の配列データベースの代わりとして、PEAKSで生成したスペクトルライブラリを使用します。
プリカーサーのm/z、電荷、アノテーションされたスペクトル情報に加え、予測の保持時間とイオン移動度のデータを使用して感度を向上させます。特にDIAデータの処理時間の短縮に有効です。
PEAKS 11では、DIA、FAIMS DIA、SWATH、diaPASEFに対してスペクトルライブラリサーチが可能です。
スペクトルライブラリの可視化・評価『PEAKS Library Viewer』
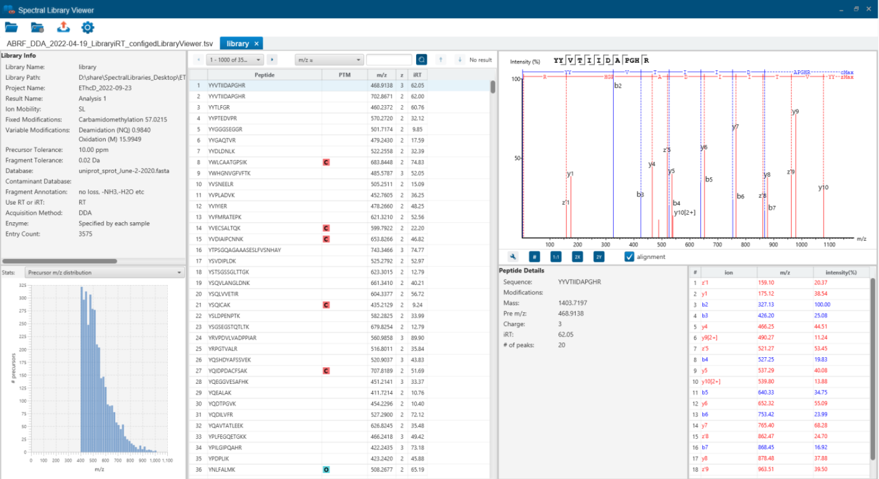
PEAKS Library Viewer では、外部もしくはPEAKS内で作成したスペクトルライブラリを簡単に可視化し、ライブラリ情報の評価と編集を行うことが可能です。
品質評価およびバリデーションのための統計解析機能を備えています。
PEAKS StudioまたはPEAKS Online から生成されたテキスト形式のライブラリ、およびSpectronaut、OpenMSから生成されたライブラリの読み込みに対応しています。
DeepNovoに基づくimmunopeptidomics用ワークフロー
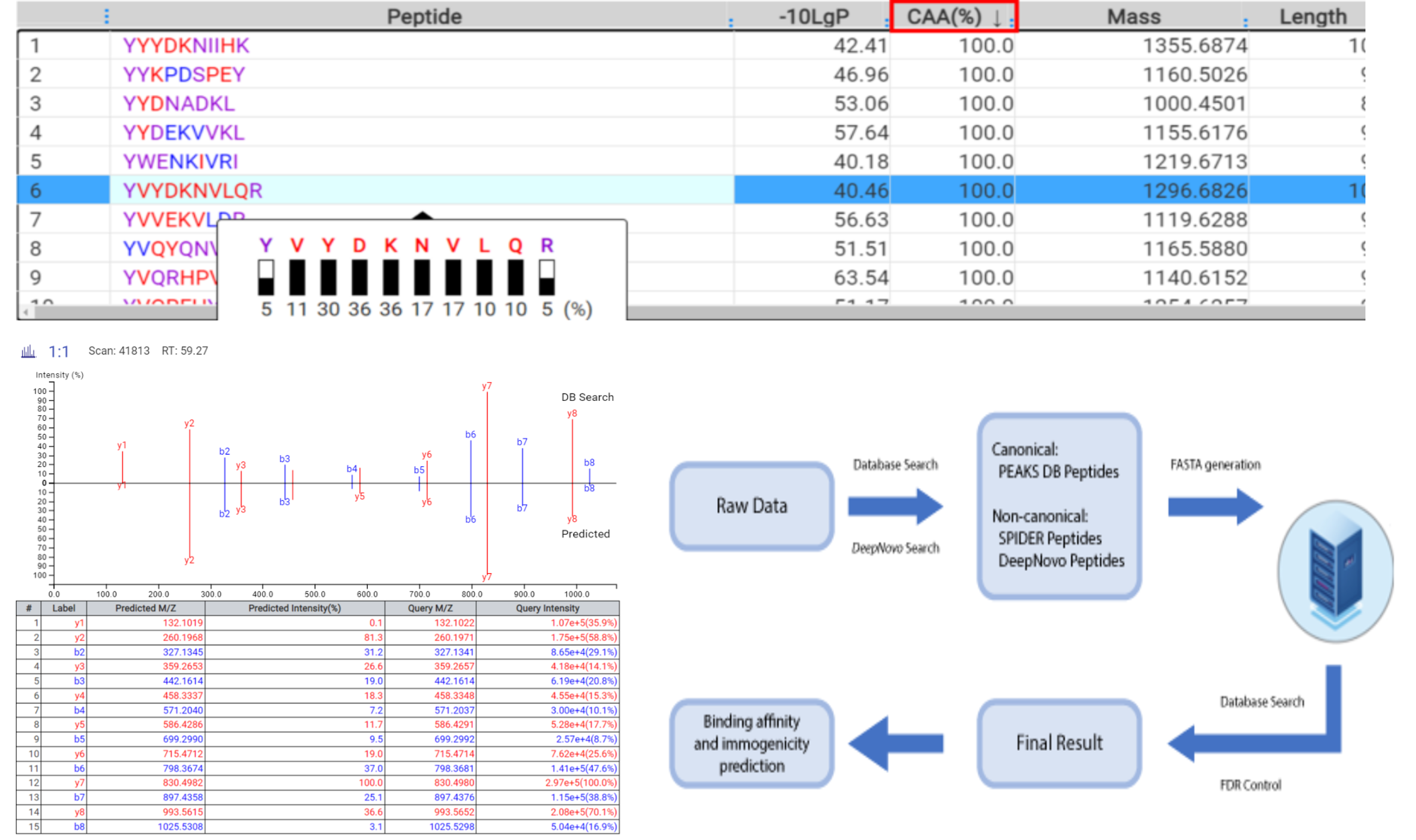
Database Search、de novo sequence、変異ペプチドの同定を組み合わせた、ペプチドミクスデータに特化したワークフローが実装されました。ペプチドミクスデータセットを用いてDeepNovoディープラーニングモデルを学習させることで、ペプチド同定の感度と精度の大幅な向上が期待され、より正確なFalse Discovery rateの推定が可能になります
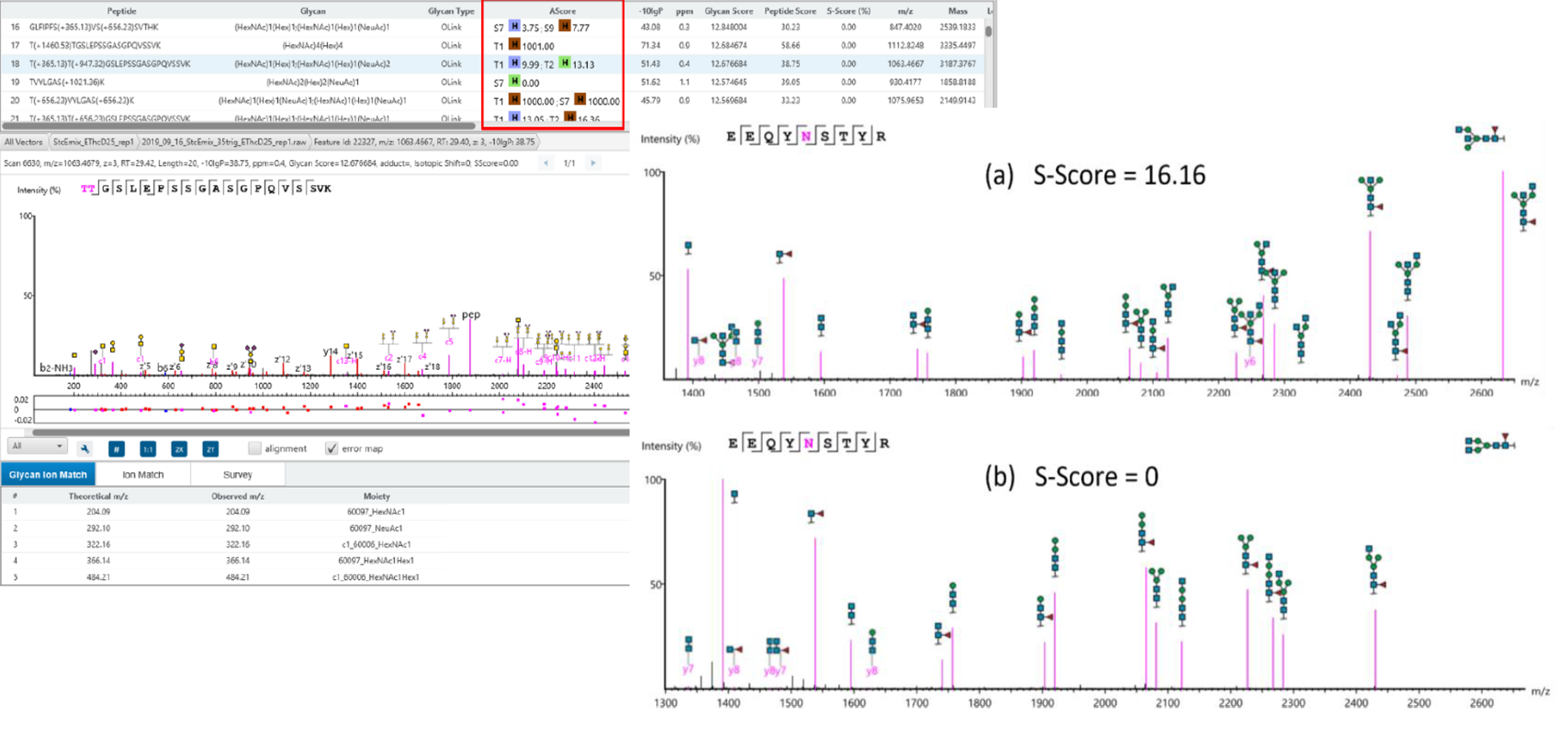
糖鎖解析機能『Glycan Search』
Glycan Searchでは、糖鎖部位の局在と糖鎖構造に対する理解を深めるために、高感度かつ高精度なデータ解析ツールです。
- LC-MS/MSデータを用いてサンプル中の糖タンパク質をプロファイリング
- N-およびO-結合型糖鎖の同定と特性解析を容易にします
PEKAS オプション
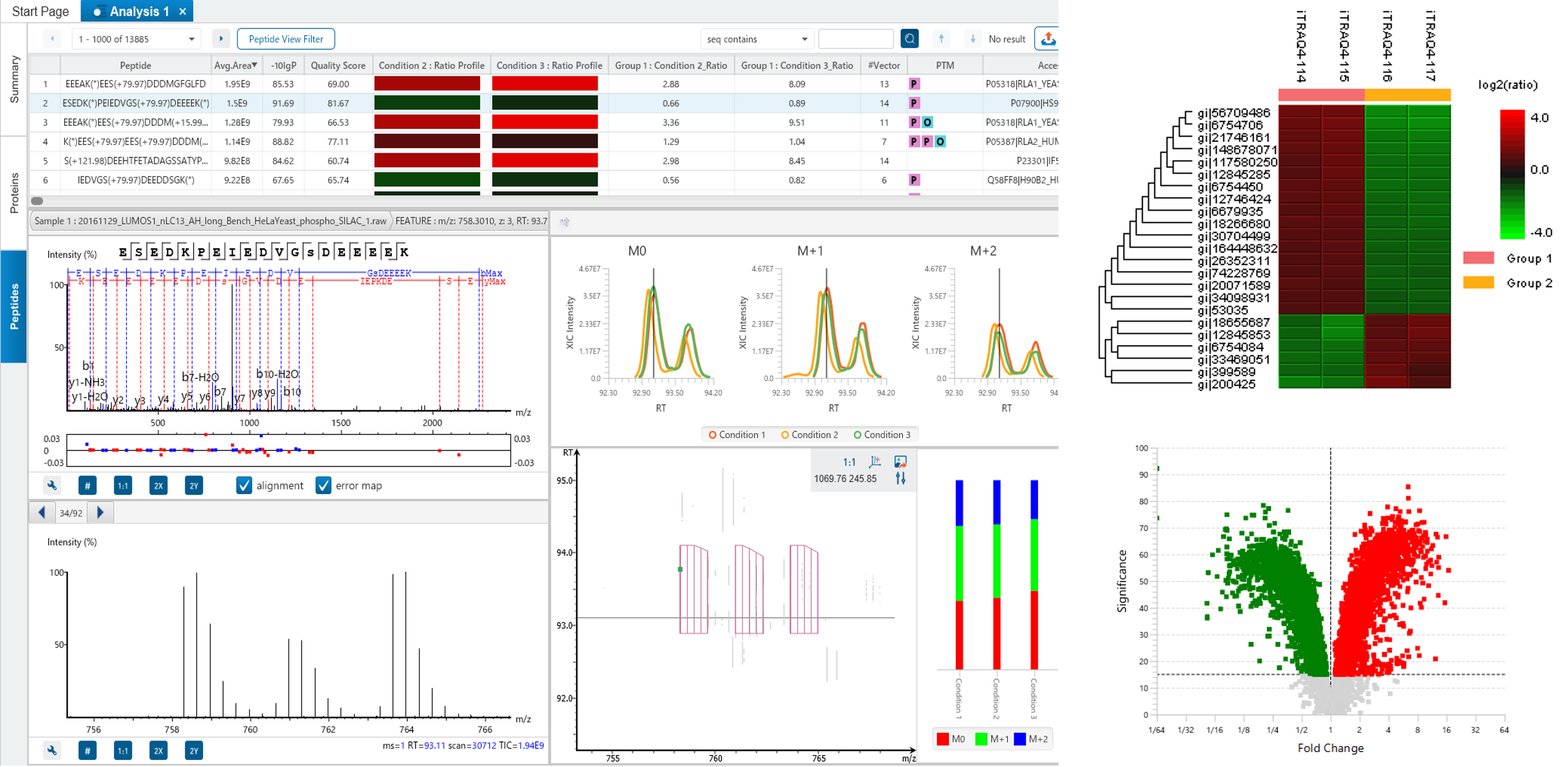
定量解析モジュール 『PEAKS Q』
PEAKS Qでは、質量分析を用いた定量解析の主な3つのプロトコルに対応しています。
- MS : 1 つのデータセット内でプリカーサーに関して得られたイオンクロマトグラム(XICs) の相対的な強度に基づく最も一般的な定量化プロトコル (例)ICAT, SILAC など
- MS/MS : MS/MS スペクトル内における固定された m/z 値のフラグメントピークの相対的な強度に基づく定量化プロトコル (例)iTRAQ, タンデムマスタグ(TMTs) など
- Label Free: 質量と溶出時間を使ってアラインメントされた複数のデータセッにおけるプリカーサーに関して得られたイオンクロマトグラム(XICs) の相対的な強度に基づく定量化プロトコル
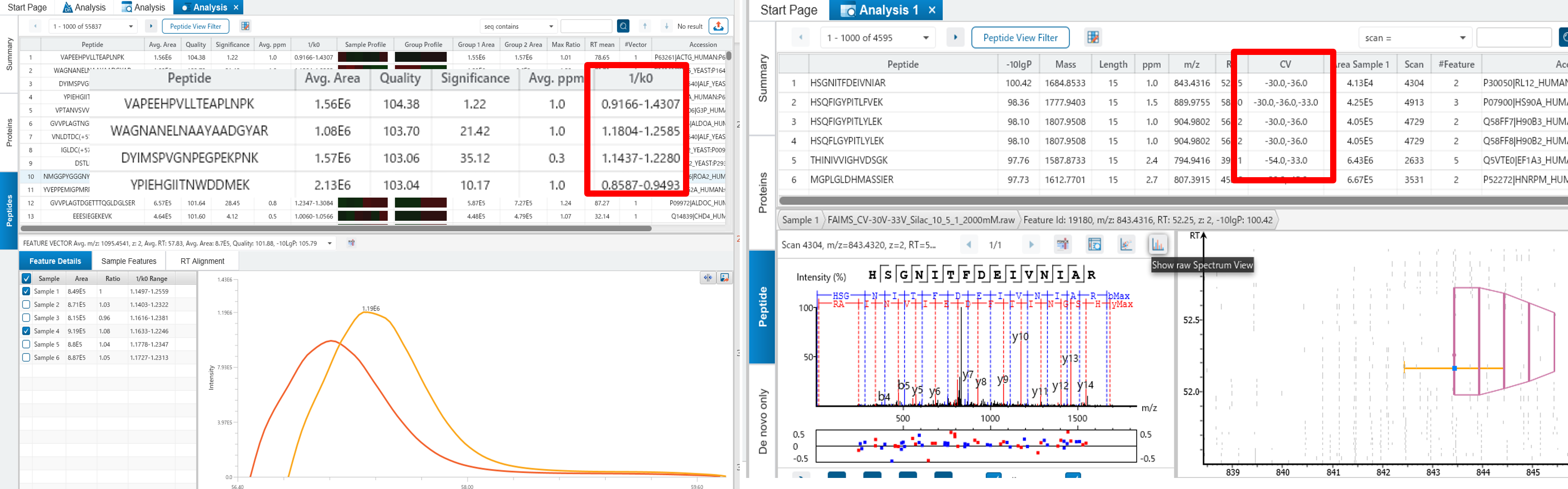
イオンモビリティデータ専用モジュール 『PEAKS IM』
PEAKS IMでは、Bruker社 timsTOF Pro / Thermo Fisher Scientific社 FAIMS / Waters社 HDMSe データで検出される三次元構造の情報を取り込む事が可能です。
- tdf/tdf_binファイル読み込み対応
- signal/featureのヒートマップ表示機能
- 1/k0値 / CV値 の結果も確認可能
- 定量解析にも対応(PEAKS Qモジュール必要)
読み込み可能なデータフォーマット

※その他はお問い合わせください。
参考: https://www.bioinfor.com/formats/
稼働環境
OS: Windows 10,11(64bit) 日本語/英語どちらも可
CPU: Quad-core processors以上 (Intel Corei7/i9/Xeon or AMD Ryzen 7/9/threadripper processors with total 32 threads 以上推奨)
RAM: 32GB以上(64GB以上推奨)
1TB以上ディスクスペース
ライセンスタイプ
解析可能なCore数上限によって以下2タイプをご用意しております。
- Desktopライセンス:16 threads(16 core)まで計算に使用可能
- Workstationライセンス: 32 threads(32 core)まで計算に使用可能
※記載の商品名等は各社の登録商標、または商品の場合があります。
